您可以通过点击 CST 信号转导通路图的各个节点查找研究资源或产品信息。您还可以下载通路图,将其用于教育和研究。
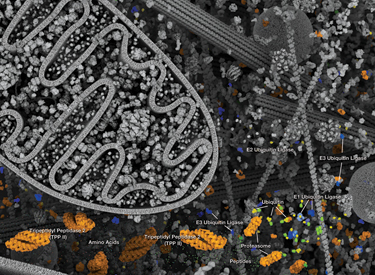
泛素-蛋白酶体系统 (UPS) 是一种细胞蛋白降解的主要方式,它是一种高度受调控的系统,可清除错误折叠或受损蛋白以及活性受信号转导通路强烈调控的蛋白。该系统在细胞增殖、转录调控、凋亡、免疫和发育中发挥核心作用。泛素是一种高度保守的含 76 个氨基酸的蛋白,能通过泛素化过程共价连接许多细胞蛋白。泛素化是一个由 3 类酶进行的依赖于 ATP 的过程。“泛素激活酶”(E1) 与泛素形成一个硫酯键。这种反应随后可使泛素结合“泛素偶联酶”(E2) 结合,之后在泛素羧基末端和底物蛋白赖氨酸残基的 ε 氨基之间形成异肽键。后一种反应需要“泛素连接酶”(E3)。在真核细胞中存在数百种 E3 连接酶;每种连接酶仅修饰一个底物蛋白亚组,从而为系统提供底物特异性。泛素化蛋白随后被靶向到 26S 蛋白酶体进行降解,或出现蛋白位置或活性变化。
泛素可作为单个单位(单泛素化)或作为支链(聚泛素化)连接底物。底物蛋白通过七种不同的泛素赖氨酸残基(Lys6、Lys11、Lys27、Lys29、Lys33、Lys48 和 Lys63)连锁至泛素。当一个泛素的赖氨酸残基连锁至另一个泛素的羧基末端甘氨酸时会形成聚泛素链。根据它们偶联所通过的泛素连接性质,聚泛素化蛋白有不同命运;K48 连接的聚泛素链主要靶向蛋白进行蛋白酶体降解,而 K63 连接的聚泛素通常调控蛋白功能、亚细胞定位和蛋白间相互作用,但这种连接有时也会导致蛋白酶体降解。
去泛素化酶 (DUB) 可去除其底物蛋白的泛素,从而逆转泛素化过程。DUB 活性可维持泛素循环,并确保细胞泛素分子集合体保持稳定。DUB 可分为 5 个亚家族:USP、UCH、OTU、MJD 和 JAMM,每个成员有特殊的组织和底物特异性。26S 蛋白酶体是一种高度富集、约 2 MDa 的复合体,可用作泛素-蛋白酶体系统的蛋白水解臂结构。它主要包含两个亚复合体:19S 调节性颗粒 (RP) 和 20S 催化核心颗粒 (CP)。CP 形成圆柱形催化室。RP 包含底座和盖。底座可展开底物并打开门控,以让未折叠的底物进入催化室。盖主要参与特异性识别泛素信号。除了 19S 帽子,其他蛋白和复合体结合 20S 圆柱的末端,并通过促进打开门控来激活它。此外,与蛋白酶体有关的 DUB 和 E3s 可重构底物锚定的聚泛素链,从而调控其对降解的敏感性。
小泛素相关修饰物 1、2 和 3(SUMO-1、-2 和 -3)和 NEDD8 是泛素样蛋白家族的成员。SUMO 和 NEDD 可使用 E1、E2、E3 偶联系统以一种类似于泛素化的方式共价连接蛋白(分别称为 sumo 化和 nedd 化)。但不像泛素化,底物蛋白的 sumo 化和 nedd 化通常不会导致降解。相反,SUMO 和 NEDD 修饰会影响亚细胞定位、蛋白功能或蛋白间相互作用。Sumo 化有许多细胞作用,包括胞核转运、转录活性调节和蛋白质稳定性。sumo 化与其他翻译后修饰(如泛素化、磷酸化和乙酰化)之间的相互作用是一个活跃的研究领域。